近日,种质创制大科学中心王翊教授课题组在生物信息学领域TOP期刊Nucleic Acids Research(《核酸研究》)上发表论文《OrthoVenn3: An Integrated Platform for Exploring and Visualizing Orthologous Data Across Genomes》。论文介绍了该团队开发的比较基因组学分析工具——OrthoVenn3。这是一个功能强大的在线分析平台,能帮助研究人员高效地识别和注释不同物种之间共享和特有的直系同源聚类,推断出物种间的进化关系、共线性区域等信息,从而促进研究者对不同物种之间的进化关系和遗传多样性的理解。

由于基因组测序的爆炸性增长,比较基因组学研究已成为生命科学研究的一个重要领域。为了便于研究者快速进行比较基因组学分析与可视化,该团队开发了全基因组比较分析在线工具——OrthoVenn。自2015年首次发布及2019年更新的OrthoVenn已累计接近1000次引用,受到60多个国家和地区研究者的青睐。最新版本的OrthoVenn3对分析功能和可视化方法等多项内容进行了更新。首先,增加了内置物种数据库的数据容量以包含更多物种并添加了基因注释信息。其次,整合了更加准确的直系同源聚类识别方法,以提高 OrthoVenn3 的准确性。第三,采用了 UpSet 可视化方法,这是一种用于在矩阵布局中可视化元素交集的工具, 使得更直观展示众多物种之间独特和共享的集群。除了这些功能之外,OrthoVenn3 还扩展了多项比较基因组学研究中常用的分析功能,包括 (i) 系统发育分析,允许根据物种的单拷贝直系同源簇推断物种之间的进化关系,(ii) 基因家族收缩和扩张分析,深入了解不同物种之间基因家族的获得或丢失,以及 (iii) 共线性分析,有助于识别基因组重排和进化的区域。
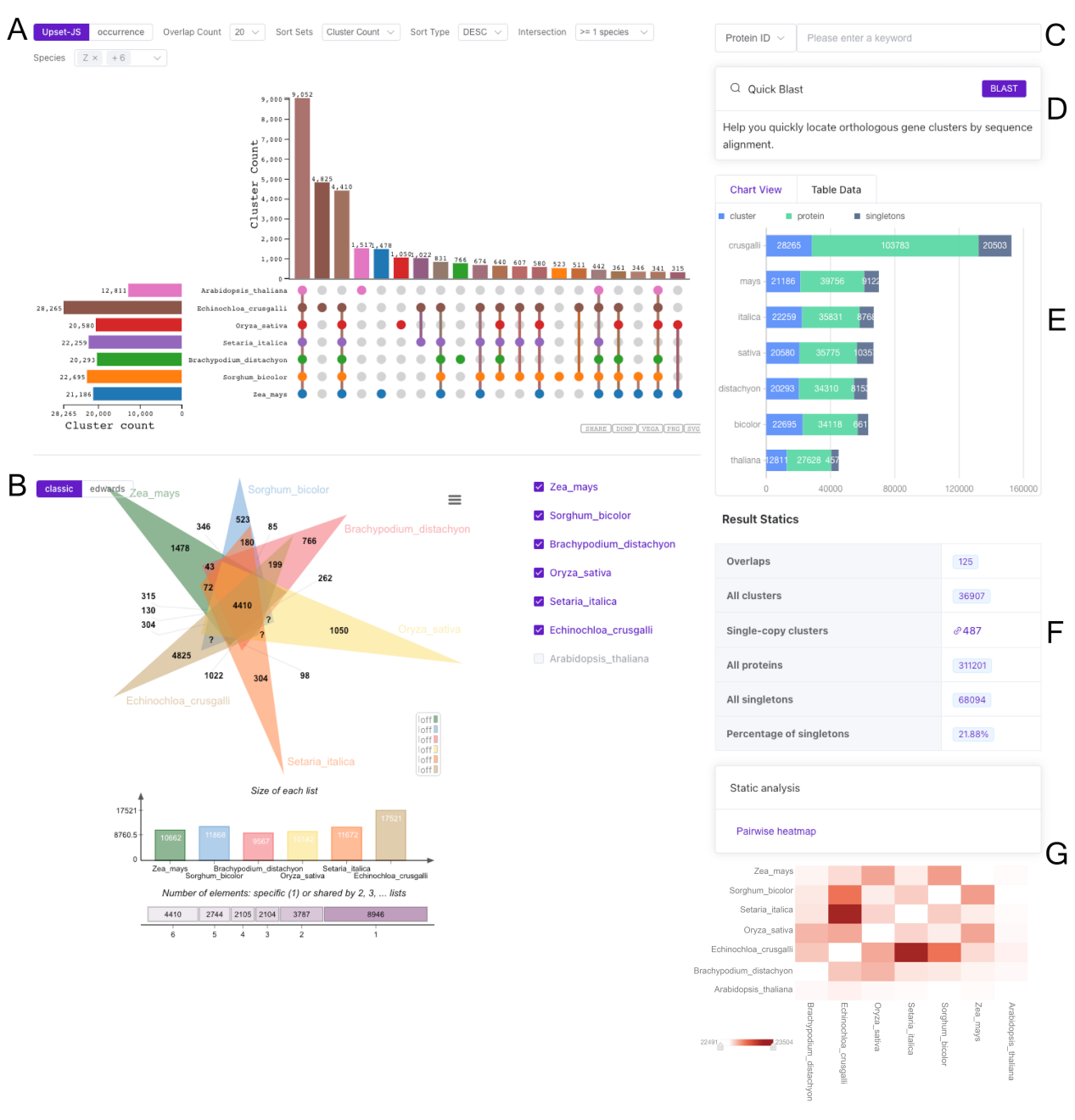
此外,OrthoVenn3还提供了基于Docker的本地版本,方便用户下载使用,本地版对不同操作系统用户提供了友好且丰富的可视化结果,无物种数量限制,可方便用户布置到任意服务器上,对比较基因组学的研究具有重要意义。
种质创制大科学中心硕士生孙家贺为论文的第一作者,王翊教授为通讯作者,中国农业大学徐凌教授为共同通讯作者。西南大学生物学研究中心大数据工程师鲁方和罗永江负责代码编写,在软件顶层设计和运算要求上发挥了重要作用,本科生别灵子也参与了数据收集工作。
该项目得到了国家自然科学基金联合基金项目、中央高校基本科研业务费专项资金等项目的资助。论文链接:https://doi.org/10.1093/nar/gkad313。