来自II型CRISPR-Cas细菌适应性免疫系统的CRISPR-Cas9已被设计为包括植物在内的多种生物体的有效基因组编辑工具。然而广泛使用的SpCas9主要识别5’-NGG-3’ PAM的特性限制了可编辑范围,特别是在A/T富集的启动子等调控序列及非蛋白编码RNA序列的有效编辑。近日,Plant Biotechnology Journal杂志在线发表了由西部(重庆)科学城种质创制大科学中心,西南大学张勇课题组及其合作团队撰写的“Expanding plant genome editing scope and profiles with CRISPR-FrCas9 systems targeting palindromic TA sites”论文。该研究针对来源于Faecalibaculum rodentium的可识别5’-NNTA-3’ 回文PAM基序的CRISPR-FrCas9植物基因组编辑新系统,构建了高活性、高特异性且识别A/T富集PAM的植物基因组编辑新工具,扩大了植物基因组编辑范围,并在此基础上进行了有效的植物蛋白编码基因敲除编辑、启动子调控编辑、microRNA敲除编辑。全文主要研究结果如下:

1.FrCas9在植物基因组编辑中的特性分析
与SpCas9相比,FrCas9的回文5’-NNTA-3’ PAM会增加sgRNA分布的密度(图1 a-c),在水稻基因组编码及非编码区,FrCas9比SpCas9可以设计更多的靶位点(图1 d)。作者测试了不同的sgRNA scaffold对于FrCas9编辑活性的影响,截短了4bp的sgRNA V02表现出最高的编辑活性(图1 e-f)。进一步,作者探究了FrCas9系统在水稻内源基因位点的编辑效率,经原生质体转化、扩增子测序结果表明,FrCas9在所选择的16个位点中均表现出了较高的编辑活性(图1 g)。同时,FrCas9在编辑点主要引起的插入多于删除(图1 h),删除主要发生在FrCas9切割位点周围(图1 i),主要产生1bp删除(图1 j),插入主要发生在PAM周围(图1 k),主要产生1bp插入(图1 l)。
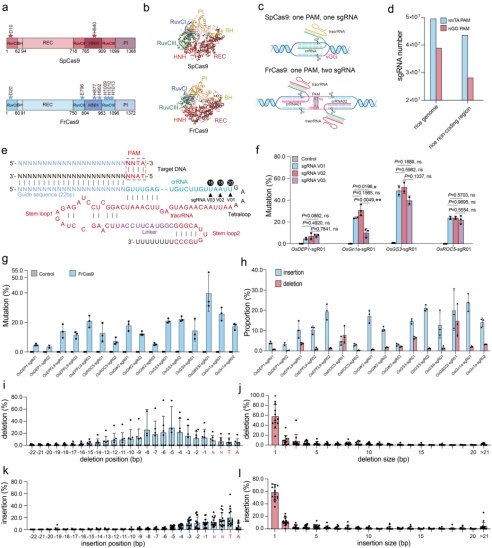
图1 CRISPR-FrCas9在植物基因组编辑中的特性研究
2.FrCas9在水稻稳定植株中的高效编辑
进一步测试FrCas9系统在水稻稳定转化植株中的编辑能力,作者在水稻T0代植株中应用FrCas9实现了有效的基因编辑,在所有测试的位点都可以获得突变的T0代植物,突变效率为10%-85.7%;在测试的6个位点中,有5个位点有双等位基因突变,双等位突变编辑效率为35%-80.7%(图2 a-b)。对FrCas9在OsGn1a和OsGS3基因编码区域产生的编辑植株进行了进一步研究,OsGn1a双等位基因突变体都显示出每穗粒数和每穗一级分枝数增加(图2 c-f);OsGS3双等位基因突变体都显示出种子长度增加,但种子宽度或种子厚度没有增加(图2 g-k)。
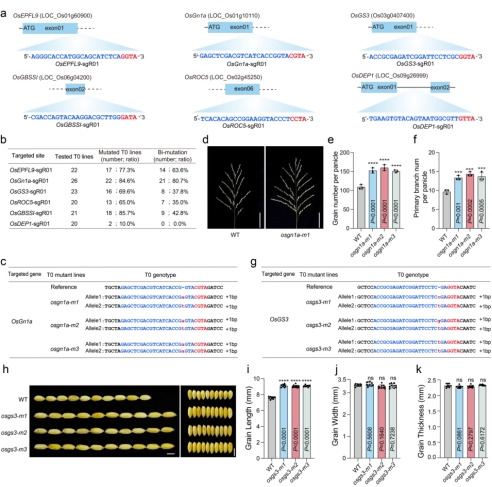
图2 CRISRP-FrCas9在水稻稳定植株中的高效基因组编辑
3.TREX2-FrCas9在不影响编辑效率的情况下实现多碱基删除编辑
FrCas9主要产生1bp的插入或者删除,这种小的InDel可以在蛋白质编码基因中产生敲除,但它们在编辑非编码基因(如微小RNA)或非编码区域(如顺式调节元件)方面不能发挥强大的作用。作者将三引物修复核酸外切酶2(TREX2)与FrCas9的N末端融合(图3 a),将TREX2-FrCas9与野生型FrCas9在水稻原生质体中的18个内源靶位点进行比较。结果显示,TREX2-FrCas9不影响编辑效率(图3 b),通过比较18个水稻内源位点的缺失谱,发现FrCas9主要产生1bp的删除,其次是2bp的删除(图3 c)。相比之下,TREX2-FrCas9产生了6bp-10bp或更长的删除,并且删除大小在16bp-20bp处达到峰值(图3 c)。此外,FrCas9在不同靶位点产生不同水平的插入和删除(图3 d),但在TREX2-FrCas9编辑的靶位点中几乎没有任何可检测的插入(图3 e)。
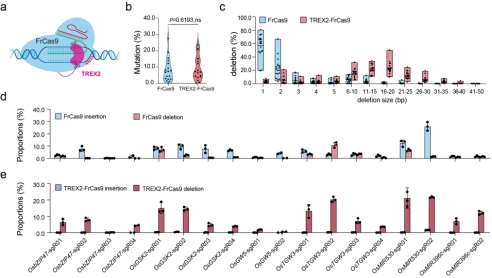
图3 TREX2-FrCas9在不影响编辑效率的情况下生成更大的删除
4.TREX2-FrCas9介导的OsMIR156j有效删除编辑
为了进一步测试了TREX2-FrCas9系统的多碱基删除能力。作者在水稻T0代植株中应用TREX2-FrCas9实现了有效的OsMIR156j编码区域多碱基删除编辑(图4 a-d),并在双等位基因删除的株系中进一步考察了OsMIR156j二级结构的变化(图4 e),OsMIR156j-5p/3p表达量的改变(图4 f),OsSPL家族部分基因的表达量的变化(图4 g),明确了TREX2-FrCas9系统作为miRNA突变体创制工具的可行性。
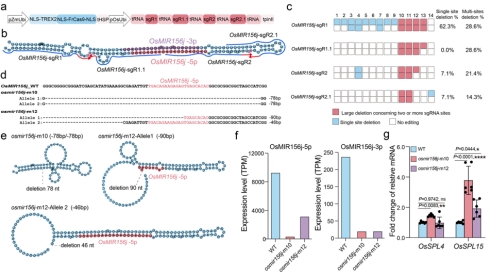
图4 TREX2-FrCas9介导的OsMIR156j有效删除编辑
5.基于FrCas9的胞嘧啶碱基编辑器及腺嘌呤碱基编辑器
基于对应于SpCas9切口酶(D10A)的FrCas9切口酶(D20A)、在哺乳动物中具有有效碱基编辑活性的FrCas9切口酶(E796A)、不同类型的胞嘧啶脱氨酶,作者在水稻中开发了不同版本的胞嘧啶碱基编辑器,实验结果表明FrCas9 CBE V2.1可以在水稻中实现高效的C-to-T碱基编辑(图5)。
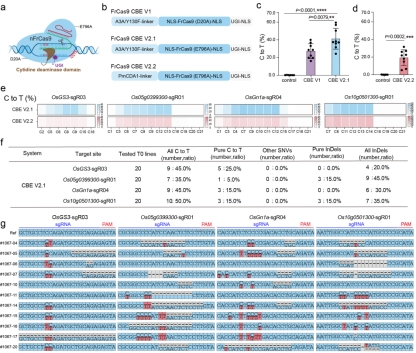
图5 基于FrCas9的胞嘧啶碱基编辑器提供有效的C-to-T碱基编辑
基于FrCas9切口酶(E796A)及腺嘌呤脱氨酶,作者在水稻中开发了腺嘌呤碱基编辑器,实验结果表明FrCas9 ABE可以在水稻中实现有效的A-to-G碱基编辑(图6)。
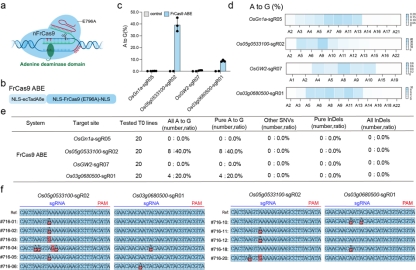
图6 基于FrCas9的腺嘌呤碱基编辑器提供有效的A-to-G碱基编辑
6.FrCas9编辑器的全基因组脱靶分析
运用WGS全基因组评估水稻FrCas9及TREX2-FrCas9系统的脱靶效应,选择18个水稻独立单株进行全基因组测序及分析(图7 a-b)。结果表明:与对照植物相比,FrCas9系统的InDel和SNV数量没有显著差异(图7 c-d);TREX2-FrCas9系统的InDel数量没有显著差异,SNV数量显著增加且主要为C:G>T:A和A:T>T:A(图7 c-f),所有SNV在基因组上随机分布(图7 g)。对所有植株的sgRNA依赖性脱靶位点进行检测,结果表明,这些sgRNA依赖性脱靶位点均没有发生突变(图7 h)。
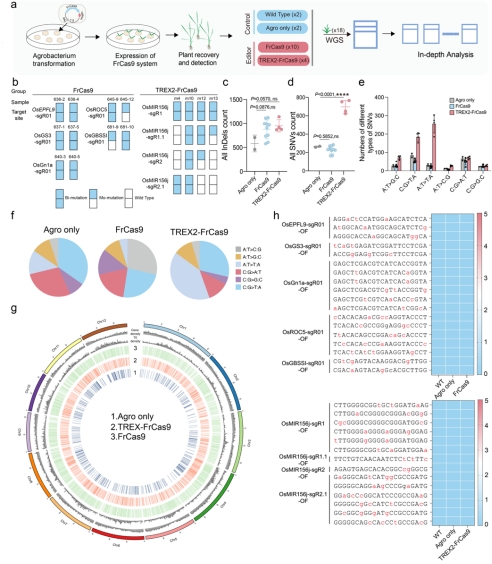
图7 FrCas9编辑器的全基因组脱靶分析
电子科技大学博士生何瑶、西南大学博士生韩阳朔为论文共同第一作者。西部(重庆)科学城种质创制大学中心,西南大学生命科学学院张勇教授、扬州大学张韬教授及马里兰大学Yiping Qi教授为论文共同通讯作者。该研究受STI 2030重大项目、国家自然科学基金面上项目等资助。
论文链接:https://onlinelibrary.wiley.com/doi/10.1111/pbi.14363