随着CRISPR-Cas核酸酶技术的飞速发展,以SpCas9为代表的基因组编辑工具已被广泛应用于植物功能基因组学研究与作物分子育种应用。经典的SpCas9编辑系统诱导DNA双链断裂(DSB),并利用细胞内源的非同源末端连接(NHEJ)途径进行修复,通常产生1 bp–2 bp碱基插入或删除(indels)为主的序列突变。对于蛋白质编码基因而言,这种碱基突变足以引起移码从而导致基因失活,可以满足基因失活敲除编辑的需求。
然而,在植物基因组中,存在大量的非蛋白编码RNA(如miRNA)及转录调控相关DNA区域(如启动子、UTR、增强子等)。这些非蛋白编码DNA区域的功能高度依赖于其特定的二级结构或关键的功能基序,1 bp–2 bp的碱基indels往往难以有效破坏miRNA结构及改变调控元件强度。虽然Cas12a编辑系统能够产生较大的碱基缺失编辑事件,是有效的非蛋白编码DNA区域编辑的有效工具,但其5’-TTTV-3’ PAM较复杂、低温编辑活性下降、多物种(特别是双子叶植物)编辑系统开发滞后,使得其应用范围受到较大限制。因此,开发一种既能高效产生多碱基删除(MNDs),又能兼顾广泛靶向范围的通用型Cas编辑系统,日渐成为针对植物非蛋白编码DNA区域进行功能解析及分子育种的迫切需求、具重要意义。

近日,西部(重庆)科学城种质创制大科学中心张勇教授课题组在Journal of Integrative Plant Biology(JIPB)期刊上,发表了题为“Enhanced exonuclease-Cas9 systems promote multiple nucleotide deletions with higher efficiency and broader targeting scope in plants”的研究论文。该研究通过引入多类型外切核酸酶及单链DNA结合结构域(DBD),成功开发了一系列多碱基删除Cas9系统(Multiple Nucleotide Deletion-Cas9, MND-Cas9),显著提升了植物基因组多碱基删除的效率和范围,为植物基因组非蛋白编码DNA区域功能解析及分子育种提供了有力工具。全文研究工作概述如下:
1、筛选高效外切酶,构建MND-Cas9 V1编辑系统
研究者基于Cas9在切割DNA后会释放非靶标链3’端的切割特性,提出通过融合单链DNA外切酶来降解释放末端,从而诱导产生大片段删除的假设。实验筛选了RecJ、T5、SbcB、Trex2四种不同特性外切酶,分别与SpCas9的N端或C端融合进行测试。结果显示,融合位置对编辑效率有显著影响,N端融合普遍优于C端。更重要的是,只有融合了3’→5’方向外切酶的SbcB-Cas9和Trex2-Cas9(即MND-Cas9 V1)显著改变了突变谱。在水稻原生质体测试中,MND-Cas9 V1系统产生的删除片段大小主要集中在6 bp–12 bp,且编辑跨度从PAM上游的4 bp–5 bp显著扩展至4 bp–15 bp。
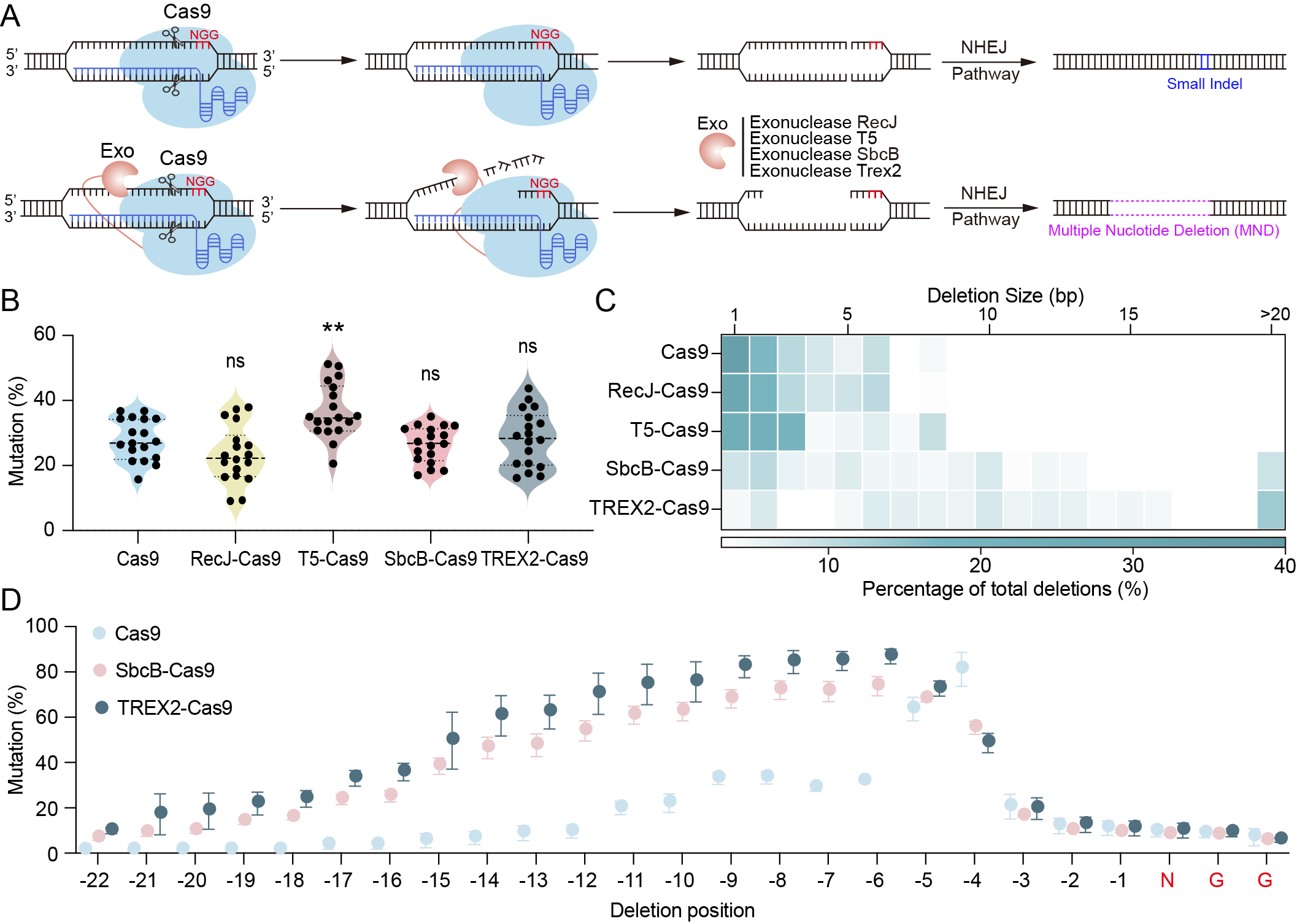
图1 MND-Cas9 V1系统设计及其在水稻中的编辑性能筛选
2、引入DBD模块优化架构,构建增强型MND-Cas9 V2系统
为了进一步提升多碱基删除的效率和一致性,研究者在Cas9与外切酶之间引入了单链DNA结合结构域(DBD),构建了MND-Cas9 V2。设计初衷是利用DBD稳定被置换的非靶标链,从而为外切酶提供更稳定的反应底物和更长的作用窗口。在水稻内源位点的评估显示,MND-Cas9 V2(SbcB-DBD-Cas9和Trex2-DBD-Cas9)不仅在编辑效率上显著超过了野生型Cas9,其产生的删除跨度也得到了进一步增强。SbcB-DBD-Cas9产生的删除主要集中在6 bp–14 bp,Trex2-DBD-Cas9则在5bp–11 bp,且最远删除位置可触达PAM上游18 bp。
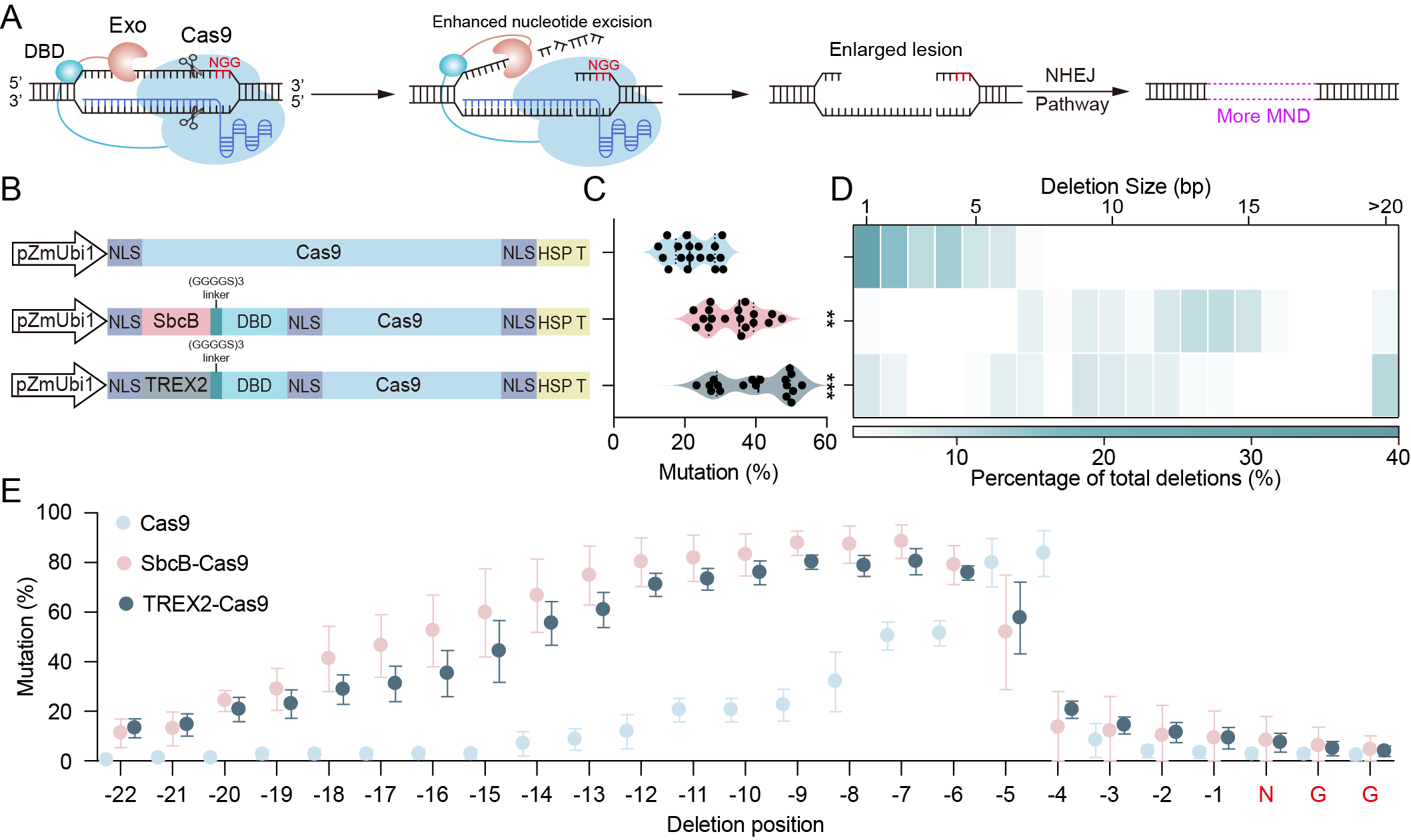
图2 MND-Cas9 V2系统优化及其突变谱分析
3、融合PAM松弛型变体,拓宽MND-Cas9系统靶向范围
针对标准SpCas9依赖NGG PAM导致的靶点选择局限性,研究者进一步将MND-Cas9 V2架构扩展至Cas9-NG和SpG等变体上,这些变体能够识别更宽泛的5’-NGN-3’ PAM。在对水稻8个具有5’-NGN-3’ PAM的内源位点进行测试后发现,MND-SpG和MND-Cas9-NG系统表现出极高的兼容性,不仅继承了Cas9变体识别多种PAM的能力,且编辑效率显著提升。
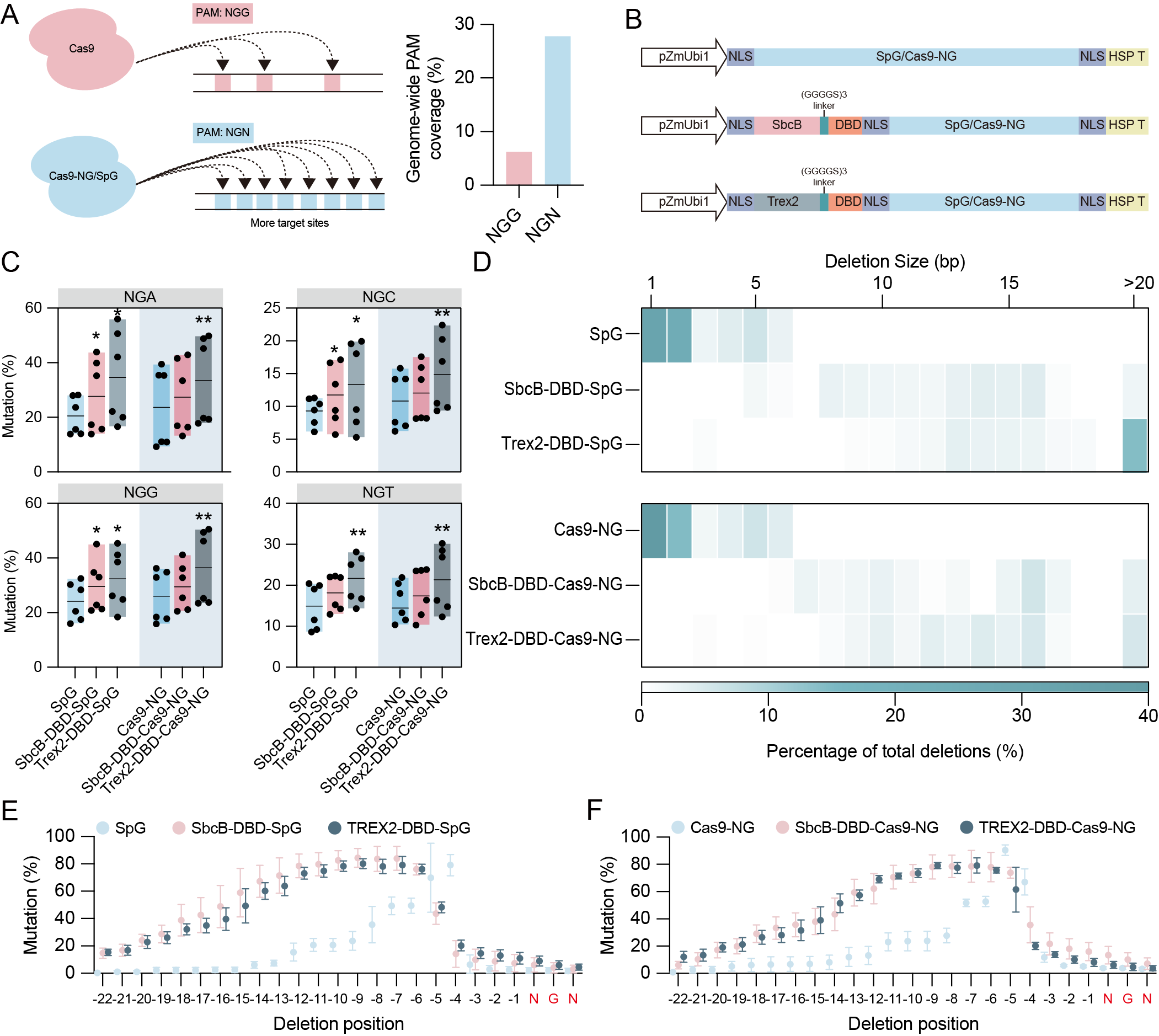
图3 基于PAM松弛型SpCas9变体的MND-Cas9 V2系统实现5’-NGN-3’ PAM位点的高效多碱基删除编辑
4、精准编辑miRNA前体结构,实现水稻粒型性状定向改良
为验证MND-Cas9系统的分子育种价值,研究者靶向了负向调节水稻籽粒大小的OsMIR530基因。由于miRNA的成熟高度依赖其前体(pre-miRNA)茎环结构的稳定性,1 bp–2 bp删除往往无法有效阻断其生物学功能。通过使用MND-Cas9 V2系统,研究者成功获得了缺失长度为7 bp–19 bp的等位突变株系。这些较大的缺失彻底破坏了OsMIR530前体的关键二级结构,导致其调控的靶基因OsPL3表达量显著上调,与野生型相比,突变体株系粒长和粒宽均显著增加。
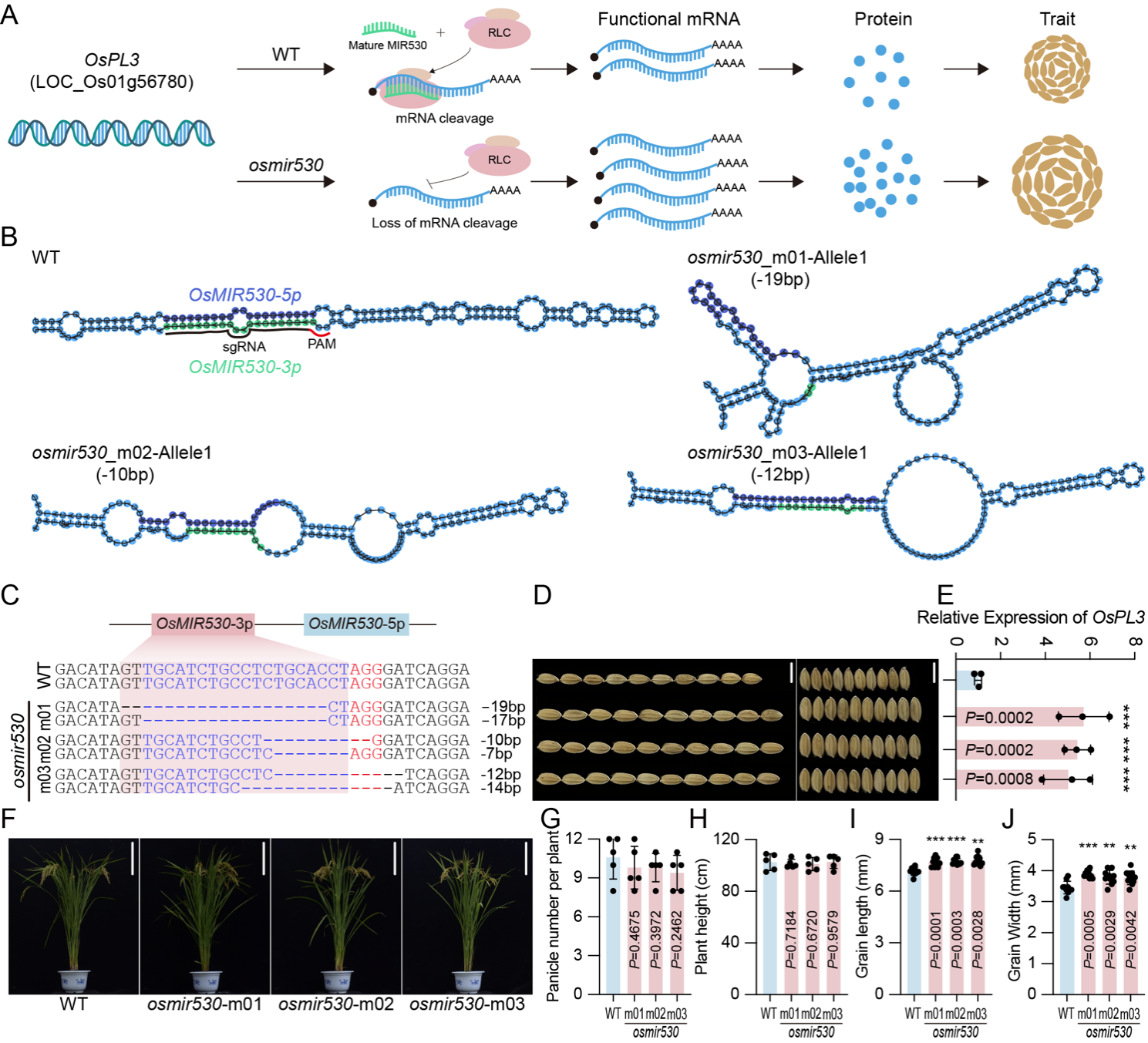
图4 应用MND-Cas9 V2系统精准编辑OsMIR530
5、编辑3' UTR调控元件,揭示顺式因子对蛋白质翻译的抑制作用
除miRNA外,研究者进一步应用MND-Cas9 V2对OsGhd2基因3’ UTR中的SMITE调控元件(研究表明:SMITE元件通过在OsGhd2基因3’ UTR形成特定的茎环结构来抑制其蛋白质翻译)进行了精准编辑。研究者采用单sgRNA局部破坏和双sgRNA大片段删除两种策略,获得了一系列OsGhd2基因3’ UTR序列发生重构的编辑突变体。实验结果显示,这些突变并未改变其mRNA水平,但双荧光报告系统验证发现,SMITE元件的缺失显著解除了翻译抑制,从而增加了蛋白积累。最终,编辑株系粒长和粒宽明显增加,为应用编辑非编码调控元件来创制数量性状变异提供了进一步典型范例。
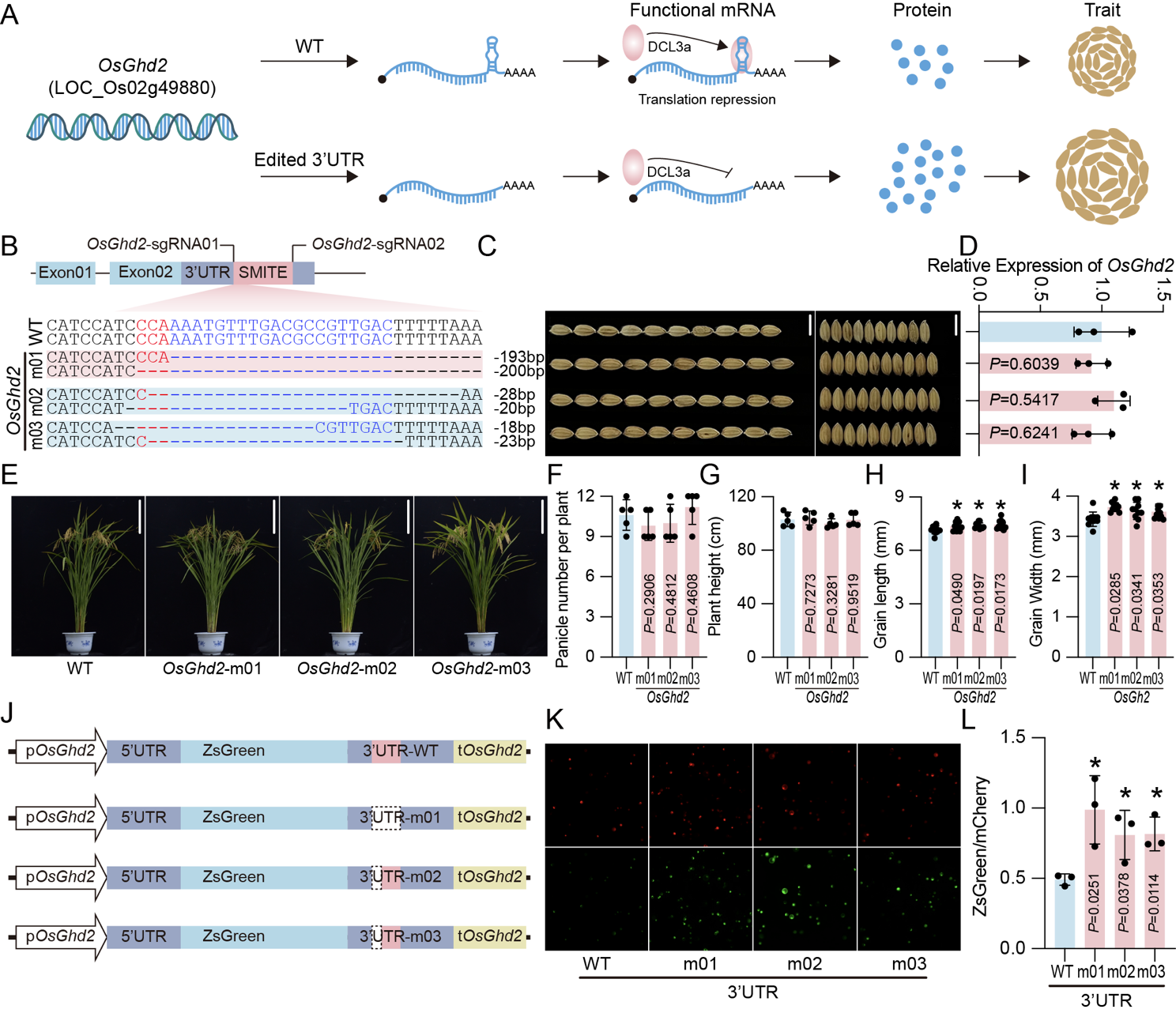
图5 OsGhd2基因3’ UTR调控元件的精准编辑
电子科技大学生命科学与技术学院博士生张瑞、西部(重庆)科学城种质创制大科学中心唐旭教授为论文共同第一作者,西部(重庆)科学城种质创制大科学中心张勇教授、马里兰大学植物科学与景观建筑系Yiping Qi教授为该研究工作共同通讯作者。研究工作得到了国家科技重大专项、国家自然科学基金等项目的资助。
论文链接:https://onlinelibrary.wiley.com/doi/10.1111/jipb.70155